Guidelines for Nomenclature of Genes, Genetic Markers, Alleles, and Mutations in Mouse and Rat
Revised: September, 2011
International Committee on Standardized Genetic Nomenclature for Mice
Chairperson: Dr. Janan T. Eppig
(e-mail:janan.eppig@jax.org)
Rat Genome and Nomenclature Committee
Rules for mouse genetic nomenclature were first published by Dunn, Gruneberg, and Snell (1940) and subsequently revised by the International Committee for Standardized Genetic Nomenclature in Mice (1963, 1973, 1981, 1989, 1996). The most recent publication of mouse nomenclature guidelines can be found in Eppig (2006). Users should be advised, however, that this web version represents the current nomenclature policies of the International Committee for Standardized Genetic Nomenclature for Mice and takes precedent over previously published versions.
Rules for rat genetic nomenclature were first published by the Committee on Rat Nomenclature in 1992 and then by Levan et al. in 1995.
In 2003, the International Committee on Standardized Genetic Nomenclature for Mice and the Rat Genome and Nomenclature Committee agreed to unify the rules and guidelines for gene, allele, and mutation nomenclature in mouse and rats. Nomenclature guidelines are now reviewed and updated annually by the two International Committees; current guidelines can be found on the MGD and RGD web sites.
http://www.informatics.jax.org/nomen/gene.shtml
잭슨사에서 만든 홈페이지에서 가져온 자료입니다 =)
첨부파일 역시 잭슨사에서 받은 것으로 플래시 파일로 공부 할 수 있어요
1 Principles of Nomenclature
1.1 Key Features
The key component of nomenclature is the gene or locus name and symbol, which identifies a unit of inheritance. Other features, such as alleles, variants and mutations, are secondary to the gene name and become associated with it. Similarly, probes or assays used to detect a gene are not primary features and should not normally be used as names.
The primary purpose of a gene or locus name and symbol is to be a unique identifier so that information about the gene in publications, databases and other forms of communication can be unambiguously associated with the correct gene. These guidelines, therefore, are intended to aid the scientific community as a whole to use genetic information.
Other, secondary, functions of nomenclature for genes are to:
- identify the gene as a member of a family, which may give further information about the gene by reference to other family members
- identify the gene as the ortholog of a gene in another mammal (usually human)
1.2 Definitions
It is important that the user understands what is being named and the principles underlying these guidelines. Section 6 presents definitions that will aid the user in distinguishing, for example, genes, loci, markers, and alleles.
1.3 Stability of Nomenclature
On the whole gene names should be stable; that is, they should not be changed over time. However there are certain circumstances where a change is desirable:
- In cases where a gene has been known only as, and named for, a mutant phenotype: when the mutated gene is identified, then the mutant name becomes the mutant allele name of the identified gene (see Section 3.1.2).
- Where a gene becomes assigned to a gene family (of paralogs), and the nomenclature of the family is established. (see Section 2.6.2).
- Where orthologous gene(s) have been identified between mouse, rat, and human, and a common symbol is adopted for all three species.
1.4 Synonyms
A gene can have several synonyms, which are names or symbols that have been applied to the gene at various times. These synonyms may be associated with the gene in databases and publications, but the established gene name and symbol should always be used as the primary identifier.
1.5 Gene symbols, proteins, and chromosome designations in publications
1.5.1 Gene and allele symbols
Gene symbols are italicized when published, as are allele symbols. Section 2 below specifies naming rules for establishing correct symbols. Help is available for determining correct gene and allele symbol assignment (nomen@jax.org) and symbols can be reserved privately pre-publication.
To distinguish between mRNA, genomic DNA, and cDNA forms within a manuscript, write the relevant prefix in parentheses before the gene symbol, for example, (mRNA) Rbp1.
1.5.2 Protein symbols
Protein designations follow the same rules as gene symbols, with the following two distinctions:
- Protein symbols use all uppercase letters.
- Protein symbols are not italicized.
1.5.3 Chromosome designations
- Use uppercase "C" when referring to a specific mouse chromosome (e.g., Chromosome 15).
- When abbreviating the word Chromosome, do not use a period (".") after the abbreviation (e.g., Chromosome 15 should abbreviated as Chr 15 and not Chr.15).
2 Symbols and Names of Genes and Loci
The prime function of a gene name is to provide a unique identifier.
The Mouse Genome Database (MGD) serves as a central repository of gene names and symbols to avoid use of the same name for different genes or use of multiple names for the same gene (http://www.informatics.jax.org). The MGD Nomenclature Committee (nomen@jax.org) provides advice and assistance in assigning new names and symbols. A web tool for proposing a new mouse locus symbol is located at the MGD site.
For the rat, these functions are carried out by RGD (http://rgd.mcw.edu) assisted by the International Rat Genome and Nomenclature Committee (RGNC). A web tool for proposing a new rat locus symbol is located at the RGD site.
2.1 Laboratory Codes
A key feature of mouse and rat nomenclature is the Laboratory Registration Code or Laboratory code, which is a code of usually three to four letters (first letter uppercase, followed by all lowercase), that identifies a particular institute, laboratory, or investigator that produced, and may hold stocks of, for example, a DNA marker, a mouse or rat strain, or were the creator of a new mutation. Laboratory codes are also used in naming chromosomal aberrations, transgenes, and genetically engineered mutations. Because Laboratory codes are key to identifying original sources, they are not assigned to "projects," but rather to the actual producer/creator individual or site. Laboratory codes can be assigned through MGD or directly by the Institute for Laboratory Animal Research (ILAR) at http://dels-old.nas.edu/ilar_n/ilarhome/register_lc.php.
Examples:
| J |
The Jackson Laboratory |
| Mit |
Massachusetts Institute of Technology |
| Leh |
Hans Lehrach |
| Kyo |
Kyoto University |
| Ztm |
Central Animal Laboratory Medical School Hannover |
2.2 Identification of New Genes
Identification of new genes in general comes in two ways; identification of a novel protein or DNA sequence or identification of a novel phenotype or trait. In the case of sequences, care should be taken in interpretation of database searches to establish novelty (for example, to distinguish between a new member of a gene family and an allele or alternative transcript of an existing family member). Novel mutant phenotypes or traits should be named according to their primary characteristic, but once the gene responsible for the phenotypic variation is identified, this gives the primary name of the gene and the mutant name becomes the name of the allele (see Section 2.3).
2.3 Gene Symbols and Names
2.3.1 Gene Symbols
Genes are given short symbols as convenient abbreviations for speaking and writing about the genes.
A gene symbol should:
- be unique within the species and should not match a symbol in another species that is not a homolog.
- be short, normally 3-5 characters, and not more than 10 characters.
- use only Roman letters and Arabic numbers.
- begin with an uppercase letter (not a number), followed by all lowercase letters / numbers (see exception below).
- not include tissue specificity or molecular weight designations.
- include punctuation only in specific special cases (see below).
- ideally have the same initial letter as the initial letter of its gene name to aid in indexing. However, letter order in a gene symbol need not follow word order in the name.
Examples:
| Plaur |
urokinase plasminogen activator receptor |
| Sta |
autosomal striping |
- be italicized in published articles. Because they may be difficult to read, depending on the browser, gene symbols are frequently not italicized when posted to a web page.
- use a common stem or root symbol when belonging to a gene family. Family member numbers or subunit designations should be placed at the end of the gene symbol.
Examples:
| Glra1 |
glycine receptor, alpha 1 subunit |
| Glra2 |
glycine receptor, alpha 2 subunit |
| Glra3 |
glycine receptor, alpha 3 subunit |
- use the same symbol whenever possible for orthologs among human, mouse and rat.
Exceptions to the rule of uppercase first letter and lowercase remaining letters in a gene or locus symbol:
- If the gene (locus) is only identified by a recessive mutant phenotype, then the symbol should begin with a lowercase letter. Once the mutant gene product is identified, the gene product is given a name and symbol and the original phenotype-based symbol and name becomes the allele symbol and name. The recessive nature of the allele is still conveyed by an initial lowercase letter.
- Within a gene symbol, Laboratory codes have an initial uppercase letter.
- When describing cross-hybridizing DNA segments, H (human) or other species code is uppercase, for example D2H11S14.
- When no information is available, other than the sequence itself, use the sequence identifier from the Mammalian Gene Collection, RIKEN, or GenBank (e.g., AF171077, 0610008A10Rik). If multiple sequence sources are available for the novel gene, preference is given first to a BC clone id (from Mammalian Gene Collection) followed by a RIKEN clone id, then the GenBank id.
Use of hyphens within the symbol should be kept to a minimum. Situations where hyphens may be used include:
- to separate related sequence and pseudogene symbols from the root
Examples:
| Hk1-rs1 |
hexokinase-1 related sequence 1 |
| Hba-ps3 |
hemoglobin alpha pseudogene 3 |
Example:
| Kit W-v |
Kit oncogene
allele name: viable dominant spotting |
2.3.2 Gene Names
Names of genes should be brief, and convey accurate information about the gene. The name should not convey detailed information about the gene or assay used; this can be associated with the gene in publications or databases. While the gene name should ideally be informative as to the function or nature of the gene, care should be taken to avoid putting inaccurate information in the name. For example, a "liver-specific protein" may be shown by subsequent studies to be expressed elsewhere.
A gene name should:
- be specific and brief, conveying the character or function of the gene.
- begin with a lowercase letter, unless it is a person's name or is a typically capitalized word.
Examples:
| Blr1 |
Burkitt lymphoma receptor 1 |
| Acly |
ATP citrate lyase |
- use American spelling.
- not contain punctuation, except where necessary to separate the main part of the name from modifiers.
Examples:
| Acp1 |
acid phosphatase 1, soluble |
| Pigq |
phosphatidylinositol glycan, class Q |
- include the name of the species from which the ortholog/homolog name was derived at the end of the name in parentheses only when that name is not in common usage.
Examples:
| Shh |
sonic hedgehog
[commonly used, does not include species name] |
| Fjx1 |
four jointed box 1 (Drosophila)
[name includes species derivative] |
- not include the word mouse (for a mouse gene name) or the word rat (for a rat gene name).
- follow the conventions of the established gene family if it is a recognizable member of that family by sequence comparison, structure (motifs/domains), and/or function.
- not contain potentially misleading information that may be experiment or assay specific, such as "kidney-specific" or "59 kDa."
2.4 Structural Genes, Splice Variants, and Promoters
Ultimately, the majority of gene names will be for structural genes that encode protein. The gene should as far as possible be given the same name as the protein, whenever the protein is identified. If the gene is recognizable by sequence comparison as a member of an established gene family, it should be named accordingly (see Section 2.6).
2.4.1 Alternative Transcripts
Alternative transcripts that originate from the same gene are not normally given different gene symbols and names. To refer to specific splice forms of a gene, the following format should be used (gene symbol, followed by underscore, followed by sequence accession ID): Genesymbol_accID
Example:
| Gene |
|
Mttp |
microsomal triglyceride transfer protein |
| Splice variant |
|
Mttp_EU553486 |
microsomal triglyceride transfer protein splice variant defined by transcript sequence EU553486 |
Using the sequence accession ID provides an unambiguous and precise definition to the splice variant.
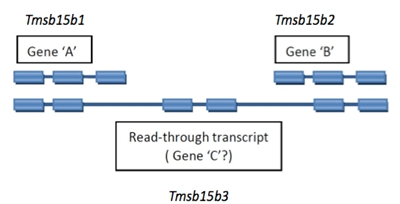
2.4.2 Read-through Transcripts
A read-through transcript is a multi-exon transcript that shares one of more exons with non-overlapping shorter transcripts that are considered to represent products of distinct loci. This is usually readily recognized as a distinct pattern, not to be confused with simple alternate splicing for a locus.
Read-through transcript genes should be named with a unique symbol and name. An example is diagrammed below.
2.4.3 Antisense and Opposite Strand Genes
Transcripts from the opposite strand that overlap another gene, or a transcript that is derived principally from the introns of another gene, or one that uses an alternative reading frame to another gene (and does not use the existing frame to a significant extent) should be given a different name.
A gene of unknown function, encoded at the same genomic locus (with overlapping exons) as another gene should have its own symbol. If the new gene regulates the first gene, it may be assigned the symbol of the first gene with the suffix “as” for antisense. The gene symbol should not be written backwards.
Example:
| Igf2as |
insulin-like growth factor 2, antisense |
Genes of unknown function on the opposite strand, which have no proven regulatory function, should be assigned the symbol of the known gene with the suffix “os” for opposite strand.
Example:
| Dnm3os |
dynamin3, opposite strand |
2.4.4 Genes with Homologs in Other Species
To aid interspecific comparison of genetic and other information, a gene that is identifiable as a homolog of an already named gene in another species can be named as "-like" "-homolog" or "-related." (Note: this is not the same as "related sequence" which applies to related sequences within mouse or within rat.) The gene name or symbol should not include the name mouse or the abbreviation "M" for mouse or the name rat or the abbreviation "R" for rat. Where possible, genes that are recognizable orthologs of already-named human genes should be given the same name and symbol as the human gene.
2.5 Phenotype Names and Symbols
Genes named for phenotypes should aim to convey the phenotype briefly and accurately in a few words. It is accepted that the name may not cover all aspects of the phenotype; what is needed is a succinct, memorable and, most importantly, unique, name. Bear in mind that identification of a variant or mutant phenotype is recognition of an allelic form of an as-yet unidentified gene that may already have or will be given a name.
2.5.1 Lethal Phenotypes
Genes identified solely by a recessive lethal phenotype with no heterozygous effect are named for the chromosomal assignment, a serial number and the name of the laboratory of origin (from the Laboratory code).
Examples:
| l5H1 |
First lethal on Chromosome 5 at Harwell |
| l4Rn2 |
Second lethal on Chromosome 4 from laboratory of Gene Rinchik |
2.6 Gene Families
Genes that appear to be members of a family should be named as family members. Evidence of gene families comes in a variety of forms, e.g., from a probe detecting multiple bands on a Southern blot, but is principally based on sequence comparisons.
2.6.1 Families Identified by Hybridization
Historically, many gene families have been identified as fragments detected by hybridization to the same probe but which map to different loci. These family members may be functional genes or pseudogenes. The loci can be named "related sequence" of the founder gene with a serial number (symbol -rs1, -rs2, and so on).
Example:
| mouse ornithine decarboxylase-related sequences 1 to 21. |
Odc-rs1 to Odc-rs21 |
If the founder or functional gene can not be identified, initially all the fragments are named "related sequence" until it is identified; then that particular "-rs" is dropped, without renumbering. If there is evidence that any loci are pseudogenes, they should be named as such and given serial numbers as in Section 2.6.2.
Once sequence evidence is accumulated on functional family members (which may or may not have been previously identified as members) a systematic naming scheme should be applied to the family as in Section 2.6.2.
2.6.2 Families Identified by Sequence Comparison
Sequencing can identify genes that are clearly members of a family (paralogs). Where possible, members of the family should be named and symbolized using the same stem followed by a serial number. The same family members in different mammalian species (orthologs) should, wherever possible, be given the same name and symbol. Pseudogenes should be suffixed by -ps and a serial number if there are multiple pseudogenes. Note that the numbering of pseudogenes among species is independent and no relationship should be implied among mouse, rat, or human pseudogenes based on their serial numbering.
Examples:
| In mouse, phosphoglycerate kinase 1, pseudogenes 1 to 7, Pgk1-ps1 to Pgk1-ps7 |
| In rat, calmodulin pseudogene 1, Calm-ps1 |
Numerous gene families have been recognized and given systematic nomenclature. Information on these families can be found at family-specific web sites, some of which are linked from MGD and RGD or RatMap. Names and symbols of new members of these families should follow the rules of the particular family and ideally be assigned in consultation with the curator of that family. Nomenclature schemes and curation of new families benefit from examination of existing models.
2.7 ESTs
Expressed Sequence Tags (ESTs) differ from other expressed sequences in that they are short, single pass sequences that are often convenient for PCR amplification from genomic DNA. ESTs that clearly derive from a known gene should be considered simply as an assay (marker) for that known gene. When anonymous ESTs are mapped onto genetic or physical maps, their designations should be symbolized using their sequence database accession number.
2.8 Anonymous DNA Segments
Only anonymous DNA segments that are mapped should be given systematic names and symbols.
2.8.1 Mapped DNA Segments
Anonymous DNA segments are named and symbolized according to the laboratory identifying or mapping the segment as "DNA segment, chromosome N, Lab Name" and a serial number, where N is the chromosomal assignment (1-19, X, Y in the mouse and 1-20, X, Y in the rat) and is symbolized as DNLabcode#.
Examples:
| D8Mit17 |
the 17th locus mapped to mouse Chromosome 8 by M.I.T. |
| D1Arb27 |
the 27th locus mapped to rat Chromosome 1 at the Arthritis and Rheumatism Branch, NIAMS. |
The same convention is applied to DNA segments that are variant loci within known genes.
Examples:
| D4Mit17 |
an SSLP within the mouse Orm1 gene |
| D20Wox37 |
an SSLP within the rat Tnf gene |
Mouse or rat DNA segments that are detected by cross-hybridization to human segments are given the human name with "chromosome N, cross-hybridizing to human DNA segment" inserted between DNA segment and the human segment code (see symbols). The same applies for rat DNA segments detected by cross-hybridization to mouse segments (or vice versa).
Examples:
| D16H21S56 |
Mouse DNA segment on Chr 16 that cross-hybridizes with a DNA segment D21S56 from human Chr 21. |
| D1M7Mit236 |
Rat DNA segment on Chr 1 that cross-hybridizes with a DNA segment D7Mit236 from mouse Chr 7 |
2.8.2 STSs Used in Physical Mapping
When physical maps are assembled (YAC or BAC contigs, for example) many markers may be placed on the map in the form of Sequence Tagged Sites (STSs). These might be clone end-fragments, inter-repeat sequence PCR products, or random sequences from within clones. These markers serve to validate the contigs and appear on the maps, but their further utility may be limited. It is not necessary to give them names or symbols other than those assigned by the laboratory that produced and used them. If the STSs are used more widely, they should be assigned anonymous DNA segment names ("D-numbers").
2.9 Gene Trap Loci
Gene trap experiments in embryonic stem (ES) cells produce cell lines in which integration into a putative gene is selected by virtue of its expression in ES cells. The trapped gene is usually (though not necessarily) mutated by the integration. The site of integration can be characterized by a number of means, including cloning or extension of cDNA products. The loci of integration of a series of gene trap lines, once characterized as potentially unique, can be named and symbolized as members of a series, using the prefix Gt (for gene trap), followed by a vector designation in parentheses, a serial number assigned by the laboratory characterizing the locus, and the laboratory ILAR code. For example, the 26th gene "trapped" by the ROSA vector in the laboratory of Phillip Soriano (Sor) is symbolized as:
A gene trap designation becomes an allele of the gene into which it was inserted, once that gene is identified. For example, Gt(ST629)Byg is known to disrupt the netrin 1 (Ntn1) gene; thus the full allele designation for this gene trap mutation is Ntn1Gt(ST629)Byg. See also the examples of gene trap mutations in Section 3.5.2.
2.10 Quantitative Trait Loci, Resistance Genes, and Immune Response Genes
Differences between inbred strains and the phenotype of offspring of crosses between strains provide evidence for the existence of genes affecting disease resistance, immune response, and many other quantitative traits (quantitative trait loci, QTL). Evidence for QTL is generally obtained through extensive genetic crossing and analysis that may uncover many genetic elements contributing to a phenotypic trait. Generally, the number and effects of QTL can only be deduced following experiments to map them. QTL should not be named until such mapping experiments have been performed.
2.10.1 Names and Symbols of QTL
Names and symbols for QTL should be brief and descriptive and reflect the trait or phenotype measured. Those QTL affecting the same trait should be given the same stem and serially numbered. The series is separate for mouse and rat and no homology should be implied by the serial numbers.
Some historically named QTL carry the name of the disease with which they are associated; these names are maintained; but newly identified QTL should be named for the measured trait and not a disease. The suffix "q" may be used optionally as the final letter preceding the serial number in QTL symbols.
Naming and symbolizing QTL follow the same conventions as for naming and symbolizing genes (Section 2.3). Specifically for a QTL, its name should include:
- a root name describing the measured trait
- the designation QTL (recommended)
- a serial number
Examples:
| in mouse |
Cafq1 |
caffeine metabolism QTL 1 |
| Cafq2 |
caffeine metabolism QTL 2 |
| Cafq3 |
caffeine metabolism QTL 3 |
| in rat |
Kidm1 |
kidney mass QTL 1 |
| Kidm2 |
kidney mass QTL 2 |
| Kidm3 |
kidney mass QTL 3 |
To obtain the next available serial number for a new QTL with an already established root name, e.g., the next in the series of "liver weight QTL" in mouse (Lwq#) or the next in series of "blood pressure QTL" for rat (Bp#), users should submit their QTL on the "proposing a new locus symbol" form at MGD (for mouse) or RGD (for rat). Note that examining the database content for a QTL is not sufficient, as a laboratory may have a QTL designation reserved and private, pending publication.
2.10.2 Defining uniqueness in QTL
Specific circumstances for naming independent QTL include:
- Independent experiments study the same trait and map that trait to the same chromosomal region
Because QTL are detected in the context of specific strain combinations in specific crosses and generally in different laboratories using different assays, each experimentally detecting QTL will be given a unique symbol/name even when the trait measured and region defined is superficially the same as that of an existing QTL.
Example: In mouse, Obq1 (obesity QTL 1) was identified and mapped to Chromosome 7 in a cross between strains 129/Sv and EL/Suz. Another obesity QTL was also mapped to Chromosome 7, but because it involved distinct strains (NZO and SM), it was given a different QTL designation, Obq15.
- A chromosomal region containing many measured "traits"
If multiple traits are measured in a single experiment and mapped to a single chromosomal region, there may or may not be evidence that different QTL are involved. If the traits are physiologically related, the QTL name should be broad enough to represent all the measured traits or the name should reflect the trait showing the highest LOD score/p-value. Conversely, if there is clear evidence that the traits are independent, each trait will constitute a unique QTL.
Examples: In mouse, Nidd1 (non-insulin-dependent diabetes mellitus 1) was associated with related measurements of plasma insulin, non-fasted blood glucose, and body weight and given a single QTL designation.
In rats, Uae5 (urinary albumin excretion QTL 5) and Cm16 (cardiac mass QTL 16) are QTLs derived from the same experiment that map to overlapping regions of Chromosome 1. Because the measured traits are independent, different QTL designations are assigned.
2.11 Chromosomal Regions
Separate documents detail guidelines for nomenclature of chromosomes (for mouse, Rules for Nomenclature of Chromosome Aberrations are online; for rat, see Levan, et al., 1995). However, certain cytological features of normal chromosomes (such as telomeres, centromeres, and nucleolar organizers) and abnormal chromosomes (such as homogeneously-staining regions and end-points of deletions, inversions, and translocations) are genetic loci that are given names and symbols.
2.11.1 Telomeres
The functional telomere should be denoted by the symbol Tel. A DNA segment that includes the telomere repeat sequence (TTAGGG)n and which maps to a telomeric location is symbolized in four parts:
- Tel (for telomere)
- The number of the chromosome
- p or q (for the short or long arm, respectively)
- A serial number, if more than one segment is assigned to the telomere
For example, Tel4q1 telomeric sequence, Chr4, q arm 1
2.11.2 Centromeres and Pericentric Heterochromatin
The functional centromere should be denoted by the symbol Cen. Until the molecular nature of a functional mammalian centromere is defined, DNA segments that map to the centromere should be given anonymous DNA segment symbols as in Section 2.8.1.
Pericentric heterochromatin, that is cytologically visible, is given the symbol Hc#, in which # is the chromosome on which it is located.
- For example, Hc14 is the pericentric heterochromatin on Chromosome 14.
Variation in heterochromatin band size can be denoted by superscripts to the symbol.
- For example, Hc14n is normal heterochromatin; superscripts l and s would be used to denote long and short heterochromatin, respectively.
2.11.3 Nucleolus Organizers
The nucleolus organizer is a cytological structure that contains the ribosomal RNA genes. These genes are given the symbols Rnr and the number of the chromosome on which they are located.
- For example, Rnr12 is the ribosomal RNA locus on Chromosome 12.
If different Rnr loci can be genetically identified on the same chromosome, they are given serial numbers in order of identification.
- For example, Rnr19-1, Rnr19-2.
2.11.4 Homogeneously Staining Regions
Homogeneously staining regions (HSRs) are amplified internal subchromosomal bands that are identified cytologically by their Giemsa staining. A DNA segment that maps within an HSR is given a conventional DNA segment symbol, when its locus is on a normal (unamplified) chromosome. When expanded into an HSR its symbol follows the guidelines for insertions, thus becoming, for example, Is(HSR;1)1Lub.
2.11.5 Chromosomal Rearrangements
Symbols for chromosomal deletions, inversions, and translocations are given in the chromosomal nomenclature section. The end points of each of these rearrangements, however, define a locus. Where there is only a single locus on a chromosome, the chromosome anomaly symbol serves to define it. However, where an anomaly gives two loci on a single chromosome they can bedistinguished by the letters p and d for proximal and distal.
- For example, In(1)1Rk-p, In(1)1Rk-d are the proximal and distal end points of the chromosomal inversion In(1)1Rk in mouse.
2.12 Genes Residing on the Mitochondria
The mitochondria carry essential genes, among them many transfer RNA (tRNA) genes. Genes residing on the mitochondria have a prefix mt- (lowercase mt followed by a hyphen). For transfer RNAs, the symbols consist of three parts, mt-, T (for tRNA), and a single lowercase letter for the amino acid. The chromosomal designation for mitochondrial genes is Chr MT.
Examples:
| mt-Tc |
tRNA, cysteine, mitochondrial
(a tRNA gene residing on the mitochondria) |
| mt-Atp6 |
ATP synthase 6, mitochondrial
(a non-tRNA gene residing on the mitochondria) |
2.13 RNA Genes Encoded in the Nucleus
There are hundreds of loci encoding transfer RNAs (tRNA) and ribosomal RNAs (rRNA), and many are encoded in the nucleus. The following method symbolizes these nuclear-encoded RNA genes:Naming nuclear encoded transfer-RNAs
Symbols for nuclear encoded transfer-RNAs consist of four parts:
| n- |
|
lowercase n followed by a hyphen to indicate nuclear encoding |
| T |
|
uppercase T to indicate transfer-RNA |
| aa |
|
the single letter abbreviation for the amino acid |
| # |
|
serial number for this transfer-RNA |
| Example: |
| n-Ta12 |
|
nuclear encoded tRNA alanine 12 (anticodon AGC) |
Naming nuclear encoded ribosomal-RNAs
Symbols for nuclear encoded ribosomal-RNAs consist of four parts:
| n- |
|
lowercase n followed by a hyphen to indicate nuclear encoding |
| R |
|
uppercase R to indicate ribosomal-RNA |
| subunit |
|
the subunit designation |
| # |
|
serial number for this ribosomal-RNA |
| Example: |
| n-R5s104 |
|
nuclear encoded rRNA 5S 104 |
2.14 microRNAs and microRNA clusters
MicroRNAs (miRNAs) are abundant, short RNA molecules that are post-transcriptional regulators that bind to complementary sequences on target mRNA transcripts, usually resulting in translational repression or target degradation and gene silencing.
Naming microRNAs Symbols for microRNAs consist of the root symbol Mir followed by the numbering scheme tracked in the miRBase database (www.mirbase.org), a database tracking microRNAs reported for all species.
For example, mouse Mir143 (microRNA 143) is represented as mmu-mir-143 in miRBase, with the mmu signifying mouse.
Naming microRNA clusters A microRNA cluster consists of several microRNAs in immediate genome proximity. These may be given symbols and names to refer unambiguously to the entire cluster.
For a microRNA cluster, the name will consist of the root symbol Mirc (for microRNA cluster) followed by a serial number (1, 2, 3…) for the cluster. MGI (for mouse) or RGD (for rat) should be consulted for the next available cluster number when a new cluster is defined. The list of microRNAs included in each cluster will be recorded in relevant database records for the genes, knockouts, and strains.
(Note that this differs from the definition of miRBase, which simply refers to clustered miRNAs as those less than 10kb from the miRNA of interest. Thus, in miRBase clusters defined based on one miRNA may or may not overlap clusters based on another miRNA.
2.15 Enhancers, Promoters, and Regulatory Regions
Enhancers, promoters, and regulatory regions can influence multiple genes. In addition, they can be localized far away from the gene(s) that they affect. Thus, it is misleading to name them based on the gene for which regulation was first recognized.
Enhancers, promoters, and regulatory regions are to be symbolized as:
| Rr# |
regulatory region # |
| where # indicates the next number in the series. |
3 Names and Symbols for Variant and Mutant Alleles
Different alleles of a gene or locus can be distinguished by a number of methods, including DNA fragment length, protein electrophoretic mobility, or variant physiological or morphological phenotype.
All mutant alleles, whether of spontaneous or induced origin, targeted mutations, gene traps, or transgenics should be submitted to MGD (mouse) or RGD (rat) for an allele or gene accession identifier.
3.1 Mutant Phenotypes
3.1.1 Genes Known Only by Mutant Phenotypes
Where a gene is known only by mutant phenotype, the gene is given the name and symbol of the first identified mutant. Symbols of mutations conferring a recessive phenotype begin with a lowercase letter; symbols for dominant or semidominant phenotype genes begin with an uppercase letter.
Examples:
| In mouse, recessive spotting, rs; abnormal feet and tail, Aft; circling, cir |
| In rat, polydactyly-luxate, lx. |
Further (allelic) mutations at the same locus, if they have the same phenotype, are given the same name with a Laboratory code preceded by a serial number (if more than one additional allele from the same lab). In the symbol the Laboratory code is added as a superscript.
- For example, agil2J, the second new allele of mouse agitans-like identified at The Jackson Laboratory.
If a new allelic mutation of a gene known only by a mutant phenotype is caused by a transgenic insertion, the symbol of this mutation should use the symbol of the transgene as superscript (see Section 3.4.2 and Section 4).
- awgTg(GBtslenv)832Pkw; mutation of abnormal wobbly gait caused by a transgene, mouse line 832, produced in the laboratory of Paul Wong. (An abbreviated form, awgTg832Pkw can be used if the abbreviated designation is unique).
If the additional allele has a different phenotype, it may be given a different name, but when symbolized the new mutant symbol is superscripted to the original mutant symbol. Also, if a new mutation is described and named but not shown to be an allele of an existing gene until later, the original name of the new mutation can be kept. Even if the phenotype is apparently identical, the original symbol is used, with the new mutation symbol as superscript.
For example
- grey coat is an allele of recessive spotting (rs) in the mouse, and hence is symbolized rsgrc.
3.1.2 Phenotypes Due to Mutations in Structural Genes
When a spontaneous or induced mutant phenotype is subsequently found to be a mutation in a structural gene, or the gene in which the mutation has occurred is cloned, the mutation becomes an allele of that gene and the symbol for the mutant allele is formed by adding the original mutant symbol as a superscript to the new gene symbol. (The mutant symbol should retain its initial upper or lowercase letter).
- The hotfoot (ho) mutation of the mouse glutamate receptor Grid2, Grid2ho.
- The dominant white spotting (W) mutation of mouse Kit, KitW
If the original mutation has multiple alleles, when describing these alleles, their symbols become part of the superscript to the identified structural gene.
- creeper, Grid2ho-cpr.
- viable white spotting, KitW-v; sash, KitW-sh.
Even if the identified gene is novel and unnamed, it is recommended that it is nevertheless given a name and symbol different from the mutant name and symbol. This will more readily allow discrimination between mutant and wild type and between gene and phenotype.
3.1.3 Wild Type Alleles and Revertants
The wild type allele of a gene is indicated by + as superscript to the mutant symbol.
- The wild type allele of the agitans-like mutation, agil+.
- The wild type Kit locus (if necessary to distinguish from mutations), Kit+.
A revertant to wild type of a mutant phenotype locus should be indicated by the symbol + with the mutant symbol as superscript.
- Revertant to wild type at the hairless mutant locus +hr
Additional revertants are given a Laboratory Code and preceded by a serial number if more than one revertant is found in a lab. Serial numbers are independent for mouse and rat revertants and no homology is implied. If the revertant is in a gene that has been cloned, then the mutant symbol is retained as superscript to the gene symbol, and + is appended.
- Revertant to wild type of the dilute mutation of myosin Va; Myo5ad+
- Second such revertant identified at The Jackson Laboratory; Myo5ad+2J.
3.2 Variants
3.2.1 Biochemical Variants
Electrophoretic or other biochemicalvariant alleles of known structural genes are usually given lowercase letters to indicate different alleles, and in the symbol the letter becomes a superscript to the gene symbol.
- For example, glucose phosphate isomerase 1 alleles a and b; Gpi1a, Gpi1b.
3.2.2 DNA Segment Variants
Variants of DNA segments are indicated by a superscript to the symbol. The symbol is usually an abbreviation for the inbred strain in which the variant is being described. However, a particular allele may be found in several inbred strains, and, furthermore, it may be difficult to establish whether an allele in one strain is identical to one in another. The use of allele symbols for DNA segments is mainly limited to describing inheritance and haplotypes in crosses. As long as the symbols are defined in the description, users are free to use whatever allele symbol best fits their needs. In tables of genotypes, the gene symbol can be omitted and the allele abbreviation used alone.
- D11Mit19a, D11Mit19b, D11Mit19c are variant alleles of D11Mit19 in mouse.
3.2.3 Single Nucleotide Polymorphisms (SNPs)
Polymorphisms defined by SNPs may occur within or outside of a protein coding sequence.
If the SNP occurs within a gene, the SNP allele can be designated based on its dbSNP_id, followed by a hyphen and the specific nucleotide.
Examples:
| Park2rs6200232-G |
The Park2 rs6200232 SNP allele with the G variant |
| Park2rs6200232-A |
The Park2 rs6200232 SNP allele with the A variant |
If the SNP occurs outside of an identified gene, the SNP locus can be designated using the dbSNP_id as the locus symbol and the nucleotide allelic variants are then superscripted as alleles. If a gene is later discovered to include this SNP locus, the same guidelines are applicable as those used when mutant locus symbols become alleles of known genes.
Examples:
| rs6200616T |
A SNP locus with the T variant |
| rs6200616C |
A SNP locus with the C variant |
Note: If a gene Xyz is later discovered to include this SNP locus, rs620061, then the alleles listed above become Xyzrs620061-T and Xyzrs620061-C.
3.3 Variation in Quantitative Trait Loci and in Response and Resistance Genes
Variation in genes that do not give rise to a visible phenotype may be detected by assaying physiological or pathological parameters. Examples of this type of variation include levels of metabolite, immune response to antigen challenge, viral resistance, or response to drugs. Genetic variation may also produce phenotypic variation in morphology, behavior, or other observable traits that interact in a complex manner with other genes and/or with the environment.
These genes can only be identified by virtue of allelic variation. In most cases, there will not be a clear wild type; hence all alleles should be named. In most cases, the alleles should be named according to their strain of origin and symbolized by adding the strain abbreviation as superscript, although for resistance and sensitivity, variants r and s may be used. Bear in mind that resistance alleles deriving from different strains may not be the same and should be given different names and symbols.
Once the gene underlying a quantitative trait has been cloned or identified, the phenotypic name should be replaced by the name of the identified gene. The allele names and symbols should be the same as those used for the phenotype.
Examples:
| Slc11a1r |
solute carrier family 11, host resistance allele |
| Slc11a1s |
solute carrier family 11, host susceptibility allele
(the QTL originally known as BCG/Lsh resistance has been identified as Slc11a1) |
| Scc2BALB/cHeA |
colon tumor susceptibility 2, BALB/cHeA allele |
| Scc2STS/A |
colon tumor susceptibility 2, STS/A allele
(for QTL Scc2, the STS/A allele has increased tumor susceptibility vs. BALB/cHeA) |
3.4 Insertional and Induced Mutations
Mutations that are induced, targeted, or selected in structural genes are named as alleles of the structural gene.
3.4.1 Mutations of Structural Genes
Variants of structural genes that are clearly mutations, whether or not they confer a phenotype, are given the superscript m#Labcode, where # is a serial number and is followed by the Laboratory code where the mutation was found or characterized. Serial numbers are independently assigned in mouse and rat and the same assigned serial number does not imply orthology. If the mutation is known to have occurred on a particular allele, that can be specified by preceding the superscript with the allele symbol and a hyphen.
- for example, Mod1a-m1Lws is a mutation of the mouse Mod1a allele, the first found in the laboratory of Susan Lewis.
If the mutation is shown to be a deletion of all or part of the structural gene, the superscript del can be used in place of m. Note that this should be used only for deletions that encompass a single gene; larger deletions should use the chromosomal deletion nomenclature.
3.4.2 Transgenic Insertional Mutations
Mutations produced by random insertion of a transgene (not by gene targeting) are named as a mutant allele of the gene (which should be given a name and symbol if it is a novel gene), with the superscript the symbol for the transgene (see Section 3.1.1 for examples, and Section 4 for naming transgenes).
3.5 Targeted and Trapped Mutations
3.5.1 Knockout, Knockin, Conditional and Other Targeted Mutations
Mutations that are the result of gene targeting by homologous recombination in ES cells are given the symbol of the targeted gene, with a superscript consisting of three parts: the symbol tm to denote a targeted mutation, a serial number from the laboratory of origin and the Laboratory code where the mutation was produced (see Section 2.1).
- For example, Cftrtm1Unc is the first targeted mutation of the cystic fibrosis transmembrane regulator (Cftr) gene produced at the University of North Carolina.
So-called "knock in" mutations, in which all or part of the coding region of one gene is replaced by another, should be given a tm symbol and the particular details of the knock-in associated with the name in publications or databases. Where there has been a replacement of the complete coding region, the replacing gene symbol can be used parenthetically as part of the allele symbol of the replaced gene along with a Laboratory code and serial number.
- For example, En1tm1(Otx2)Wrst where the coding region of En1 was replaced by the Otx2 gene, originating from the W. Wurst laboratory.
Knock in alleles expressing a RNAi under the control of the endogenous promoter can be designated using targeted mutation or transgene mutation nomenclature, as appropriate:
Example:
When a targeting vector is used to generate multiple germline transmissible alleles, such as in the Cre-Lox system, the original knock-in of loxP would follow the regular tm designation rules. If a second heritable allele was then generated after mating with a cre transgenic mouse, it would retain the parental designation followed by a decimal point and serial number.
- Tfamtm1Lrsn and Tfamtm1.1Lrsn. In this example, Tfamtm1Lrsn designates a targeted mutation where loxP was inserted into the Tfam gene. Tfamtm1.1Lrsn designates another germline transmissible allele generated after mating with a cre transgenic mouse. Note: somatic events generated in offspring from a Tfamtm1Lrsn bearing mouse and a cre transgenic that cause disruption of Tfam in selective tissues would not be assigned nomenclature.
Other more complex forms of gene replacement, such as partial "knock-in", hit-and-run, double replacements, and loxP mediated integrations are not conveniently abbreviated and should be given a conventional tm#Labcode superscript. Details of the targeted locus should be given in associated publications and database entries.
Note that although subtle alterations made in a gene appear to lend themselves to a simple naming convention whereby the base or amino acid changes are specified, in fact these do not provide unique gene names, as such alterations, which could be made in independent labs, while bearing the same changes, may differ elsewhere in the gene.
Large-scale projects that systematically produce a large number of alleles (>1000) may include a project abbreviation in parentheses as part of the allele designation. These should retain the accepted nomenclature features of other alleles of that class. For example, a targeted allele created by Velocigene (Regeneron) in the KOMP knockout project:
Gstm3tm1(KOMP)Vlcg
Once fully designated in a publication, the allele can be abbreviated by removing the portion of the allele designation in parentheses (in this case, Gstm3tm1Vlcg), providing the symbol remains unique.
3.5.2 Endonuclease-induced Mutations
Endonuclease-induced mutations are targeted mutations generated in pluripotent or totipotent cells by an endonuclease joined to sequence-specific DNA binding domains. The mutation is introduced during homology-directed or non-homologous end-joining repair of the induced DNA break(s). Endonuclease-induced mutations are given the symbol of the mutated gene, with a superscript consisting of three parts: the symbol em to denote an endonuclease-induced mutation, a serial number from the laboratory of origin and the Laboratory code where the mutation was produced.
Example:
| Fgf1em1Mcw I |
|
the first endonuclease-induced mutation of the fibroblast growth factor 1 (Fgf1) gene produced at the Medical College of Wisconsin. |
3.5.3 Gene Trap Mutations
Gene trap mutations are symbolized in a similar way to targeted mutations. If the trapped gene is known, the symbol for the trapped allele will be similar to a targeted mutation of the same gene using the format Gt(vector content)#Labcode for the allele designation. Example:
| Akap12Gt(ble-lacZ)15Brr |
|
a gene trap allele of the Akap12 gene, where the gene trap vector contains a phleomycin resistance gene (ble) and lacZ, the 15th analyzed in the laboratory of Jacqueline Barra (Brr). |
If the trapped gene is novel, it should be given a name and a symbol, which includes the letters Gt for "gene trap," the vector in parentheses, a serial number, and Laboratory code.
- For example, a gene trapped locus (where the gene is unknown) using vector ROSA, the 26th made in P. Soriano's laboratory, is Gt(ROSA)26Sor.
For high throughput systematic gene trap pipelines, the mutant ES cell line's designation can be used in parentheses instead of the vector designation, and the serial number following the parentheses may be omitted.
Examples:
| Gt(DTM030)Byg |
for a trapped gene (at an undefined locus) in mutant ES cell line DTM030, made by BayGenomics |
| Osbpl1aGt(OST48536)Lex |
gene trap allele of the oxysterol binding protein-like 1A gene, in mutant ES cell line OST48536, made by Lexicon Genetics, Inc. |
3.5.4 Enhancer Traps
Enhancer traps are specialized transgenes. One utility of these transgenes is in creating cre driver lines. Enhancer traps of this type that are currently being created may include a minimal promoter, introns, a cre recombinase cassette (sometimes fused with another element such as ERT2), and polyA sites from different sources.
Nomenclature for these enhancer traps consists of 4 parts as follows:
| Et |
|
prefix for enhancer trap |
| cre recombinase cassette |
|
portion in parentheses...
for example, cre, icre, or cre/ERT2 (if fused with ERT2) |
| line number or serial number |
|
to designate lab trap number or serial number |
| Lab code |
|
ILAR code identifying the creator of this enhancer trap |
Examples:
| Et(icre)1642Rdav |
|
Enhancer trap 1642, Ron Davis |
| Et(cre/ERT2)2047Rdav |
|
Enhancer trap 2047, Ron Davis |
Note that the minimal promoter, poly A source, etc. are not part of the enhancer trap nomenclature. These are molecular details of the specific construct that will be captured in database records and reported with experimental results.
4 Transgenes
Any DNA that has been stably introduced into the germline of mice or rats is a transgene. Transgenes can be broken down into two categories:
- Those that are produced by homologous recombination as targeted events at particular loci.
- Those that occur by random insertion into the genome (usually by means of microinjection).
Nomenclature for targeted genes is dealt with in Section 3.5. Random insertion of a transgene in or near an endogenous gene may produce a new allele of this gene. This new allele should be named as described in Section 3.4.2. The transgene itself is a new genetic entity for which a name may be required. This section describes the guidelines for naming the inserted transgene.
It is recognized that it is not necessary, or even desirable, to name all transgenes. For example, if a number of transgenic lines are described in a publication but not all are subsequently maintained or archived, then only those that are maintained require standardized names. The following Guidelines were developed by an interspecies committee sponsored by ILAR in 1992 and modified by the Nomenclature Committee in 1999 and 2000. Transgenic symbols should be submitted to MGD or RGD/RatMap through the usual nomenclature submission form for new loci. The transgene symbol is made up of four parts:
- Tg denoting transgene.
- In parentheses, the official gene symbol of the inserted DNA, using nomenclature conventions of the species of origin.
- The laboratory's line or founder designation or a serial number (note that numbering is independent for mouse and rat series).
- The Laboratory code of the originating lab.
Note that, in contrast to gene and allele symbols, transgene symbols are not italicized as they are random insertions of foreign DNA material and are not part of the native mouse genome.
Examples:
| Tg(Zfp38)D1Htz |
a transgene containing the mouse Zfp38 gene, in line D1 reported by Nathaniel Heintz. |
| Tg(CD8)1Jwg |
a transgene containing the human CD8 gene, the first transgenic line using this construct described by the lab of Jon W. Gordon. |
| Tg(HLA-B*2705, B2M)33-3Trg |
a double transgene in rat containing the human HLA-B*2705 and B2M genes, that were co-injected, giving rise to line 33-3 by Joel D. Taurog. |
The *, as used in the last example above, indicates that the included gene is mutant.
Different transgenic constructs containing the same gene should not be differentiated in the symbol; they will use the same gene symbol in parentheses and will be distinguished by the serial number/Laboratory code. Information about the nature of the transgenic entity should be given in associated publications and database entries.
In many cases, a large number of transgenic lines are made from the same gene construct and only differ by tissue specificity of expression. The most common of these are transgenes that use reporter constructs or recombinases (e.g., GFP, lacZ, cre), where the promoter should be specified as the first part of the gene insertion designation, separated by a hyphen from the reporter or recombinase designation. The SV40 large T antigen is another example. The use of promoter designations is helpful in such cases.
Examples:
| Tg(Wnt1-LacZ)206Amc |
the LacZ transgene with a Wnt1 promoter, from mouse line 206 in the laboratory of Andrew McMahon. |
| Tg(Zp3-cre)3Mrt |
the cre transgene with a Zp3 promoter, the third transgenic mouse line from the laboratory of Gail Martin. |
In the case of a fusion gene insert, where roughly equal parts of two genes compose the construct, a forward slash separates the two genes in parentheses.
Example:
| Tg(TCF3/HLF)1Mlc |
a transgene in which the human transcription factor 3 gene and the hepatic leukemia factor gene were inserted as a fusion chimeric cDNA, the first transgenic mouse line produced by Michael L. Cleary's laboratory (Mlc). |
This scheme is to name the transgene entity only. The mouse or rat strain on which the transgene is maintained should be named separately as in the Rules and Guidelines for Nomenclature of Mouse and Rat Strains. In describing a transgenic mouse or rat strain, the strain name should precede the transgene designation.
Examples:
| C57BL/6J-Tg(CD8)1Jwg |
mouse strain C57BL/6J carrying the Tg(CD8)1Jwg transgene. |
| F344/CrlBR-Tg(HLA-B*2705, B2M)33-3Trg |
rat strain F344/CrlBR carrying the Tg(HLA-B*2705,B2M)33-3Trg double transgene. |
For BAC transgenics, the insert designation is the BAC clone and follows the same naming convention as the Clone Registry at NCBI.
Example:
| Tg(RP22-412K21)15Som |
a BAC transgene where the inserted BAC is from the RP22 BAC library, plate 412, row K, column 21. It is the 15th in the mouse made in the laboratory of Stefan Somlo (Som). |
Transgenes containing RNAi constructs can be designated minimally as:
| Tg(RNAi:geneX)#Labcode, where |
| geneX |
is the gene that is knocked down |
| # |
is the serial number of the transgene |
An expanded version of this designation is:
| Tg(Pro-yyRNAi:geneX)#Labcode, where |
| Pro- |
can be used optionally to designate the promoter |
| yy |
can be used optionally for the specific RNAi construct |
While there is the option to include significant information on vectors, promoters, etc. within the parentheses of a transgene symbol, this should be minimized for brevity and clarity. The function of a symbol is to provide a unique designation to a gene, locus, or mutation. The fine molecular detail of these loci and mutations should reside in databases such as MGD and RGD.
5 Transposon-induced Mutations and Inserts
Three types of genetic inserts are involved in creating transposon-induced mutations. Two lines, one carrying the transposable-element as a concatamer and the other carrying the transposase are mated. This causes the transposable-element to come in contact with the transposase and to be mobilized from its original site, and, when reintegrated into the genome, can cause a heritable phenotypic mutation. (c.f., Ding, et al.,2005; Bestor, 2005;Dupuy, et al., 2005). Accepted nomenclature for the transposable-element inserts, transposase transgenes, and resulting transposed insertion alleles are given below.
5.1 Transgenic Transposable Element (TE) Concatamers
The transgenic transposable element concatamers are identified with a standard prefix Tg (for transgenic) and Tn (for transposable element). The class of transposable element may be included in parentheses. The general format of the symbol is:
TgTn(transposon_class_abbreviation-vector)#Labcode
Example: TgTn(sb-T2/GT2/tTA)1Dla
The symbol consists of:
- Tg denoting transgenic
- Tn denoting transposon
- In parentheses, a lowercase abbreviation of the transposon class (in this case sb for Sleeping Beauty), followed by a hyphen and the vector designation
- The laboratory's line or founder designation or a serial number
- The Laboratory Code of the originating lab
5.2 Transposase Inserts
Transposases can be engineered into the genome via transgenesis or specific gene targeting. In these cases the relevant nomenclature for transgenes or targeted mutations is used.
For a transgene, use the standard prefix Tg (for transgene). The contents of the parentheses will usually be the promoter and the symbol for the transposase with which it is associated, separated by a hyphen. The general format of the symbol is:
Tg(promoter-transposase)#Labcode
Example: Tg(ACTB-sb10)545Abc
The symbol consists of:
- Tg denoting transgene
- In parentheses, the official gene symbol for the promoter, using the nomenclature of the species of origin, followed by a hyphen and a lowercase transposase symbol, in this case sb10 for the Sleeping Beauty 10 transposase
- The laboratory's line or founder designation or a serial number
- The Laboratory code of the originating lab.
For a targeted knock-in of the transposase, use the standard format for a targeted mutation, i.e., the symbol of the targeted gene with a superscripted allele symbol beginning with the prefix tm. The contents of the parentheses will usually be the symbol for the transposase with which it is associated. The general format of the symbol is:
Genetm#(transposase)Labcode
Example: Gt(ROSA)26Sortm1(sb11)Njen
The symbol consists of:
- The gene into which the transposase was integrated, in this case Gt(ROSA)26Sor
- In the superscript:
- tm denoting targeted mutation
- A serial number of the targeted mutation
- In parentheses, a lowercase transposase symbol, in this case sb11 for the Sleeping Beauty 11 transposase
- The Laboratory Code of the originating lab
5.3 Transposed Insertion Alleles
These alleles follow the rules for naming all other alleles. In general a transposable element concatamer marker will already be established, as above. The new allele, then, will be a superscripted form of the concatamer symbol. Note that all such alleles that are "derived from" a transposable element concatamer carry the original number with a decimal point and serial number identifying the specific allele. The general format is:
GeneTn(transposon_class_abbreviation-vector)#Labcode
Example: Car12Tn(sb-T2/GT2/tTA)1.1Dla
The symbol consists of:
The gene into which the transposable element was integrated (transposed)
In the superscript:
- Tn denoting transposon
- In parentheses, a lowercase abbreviation of the transposon class (in this case sb for Sleeping Beauty), followed by a hyphen and the vector designation
- A serial number, in which the primary number corresponds to that given to the transposable element concatamer from which it arose, followed by a decimal point and a serial number designating its number within the series of derivative insertion alleles.
- The Laboratory Code of the lab originating the transposable element line.
If a newly transposed insertion occurs in an unknown site or intergenic region, the form:
Tn(transposon_class_abbreviation-vector)#Labcode
is used to symbolize the "genomic mutation" without being superscripted to a gene symbol, similar to the way a random transgene inserted into a non-gene site is designated.
6 Definitions
The following definitions should aid the user in understanding what is being named, and in understanding the principles underlying these guidelines.
6.1 Gene
A gene is a functional unit, usually encoding a protein or RNA, whose inheritance can be followed experimentally. Inheritance is usually assayed in genetic crosses, but identification of the gene in cytogenetic or physical maps are other means of mapping the locus of a gene. The existence of a gene can also be inferred in the absence of any genetic or physical map information, such as from a cDNA sequence.
6.2 Pseudogene
A sequence that closely resembles a known functional gene, at another locus within a genome, that is non-functional as a consequence of (usually several) mutations that prevent either its transcription or translation (or both). In general, pseudogenes result from either reverse transcription of a transcript of their "normal" paralog (in which case the pseudogene typically lacks introns and includes a poly(A) tail; often called processed pseudogenes) or from recombination (in which case the pseudogene is typically a tandem duplication of its "normal" paralog).
6.3 Locus
A locus is a point in the genome, identified by a marker, which can be mapped by some means. It does not necessarily correspond to a gene; it could, for example, be an anonymous non-coding DNA segment or a cytogenetic feature. A single gene may have several loci within it (each defined by different markers) and these markers may be separated in genetic or physical mapping experiments. In such cases, it is useful to define these different loci, but normally the gene name should be used to designate the gene itself, as this usually will convey the most information.
6.4 Marker
A marker is the means by which a gene or a locus is identified. The marker is dependent on an assay, which could, for example, be identification of a mutant phenotype or presence of an enzyme activity, protein band, or DNA fragment. The assay must show genetic variation of the marker to map the locus on a genetic map but not to place it on a physical map.
6.5 Allele
The two copies of an autosomal gene or locus on the maternal and paternal chromosomes are alleles. If the two alleles are identical, the animal is homozygous at that locus. When genetically inherited variants of a gene or locus are detectable by any means, the different alleles enable genetic mapping. A single chromosome can only carry a single allele and, except in cases of duplication, deletion or trisomy, an animal carries two autosomal alleles. In particular, a transgene inserted randomly in the genome is not an allele of the endogenous locus; the condition is termed hemizygous if the transgene is present only in one of the two parental chromosome sets. By contrast, a gene modified by targeting at the endogenous locus is an allele and should be named as such.
6.6 Allelic Variant
Allelic variants are differences between alleles, detectable by any assay. For example, differences in anonymous DNA sequences can be detected as simple sequence length polymorphism (SSLP) or single nucleotide polymorphisms (SNPs). Other types of variants include differences in protein molecular weight or charge, differences in enzyme activity, or differences in single-stranded conformation (SSCP). Many allelic variants, in particular DNA variants, do not confer any external phenotype on the animal. These variants are often termed “polymorphisms” although, strictly speaking, that term applies only to variants with a frequency of more than 1% in the population.
6.7 Splice Variant or Alternative Splice
Alternative splicing of a gene results in different, normally occurring forms of mRNA defined by which exons (or parts of exons) are used. Thus one or more alternative protein products can be produced by a single allele of a gene. Among different alleles, alternative splice forms may or may not differ, depending on whether the sequence difference between the alleles affects the normal splicing mechanism and results in differences in the exon (or partial exon) usage. For example, allele A may produce mRNAs of splice form 1, 2, and 3; while allele B may produce mRNAs of splice form 1, 2, and 4; and Allele C may produce mRNAs of splice form 1, 2, and 3. In this case, each of the alleles A, B, and C by definition must differ in their DNA sequence. However, the difference between allele B versus alleles A and C must include a sequence difference that affects the splicing pattern of the gene.
6.8 Mutation
A mutation is a particular class of variant allele that usually confers a phenotypically identifiable difference to a reference "wild type" phenotype. However, in some cases, such as when homologous recombination is used to target a gene, a readily identified phenotype may not result even though the gene may be rendered non-functional. In such cases, the targeted genes are nevertheless referred to as mutant alleles.
6.9 Dominant and Recessive
Dominant and recessive refer to the nature of inheritance of phenotypes, not to genes, alleles, or mutations. A recessive phenotype is one that is only detected when both alleles have a particular variant or mutation. A dominant phenotype is detectable when only one variant allele is present. If both alleles can be simultaneously detected by an assay, then they are codominant. For example, an assay that detects variation of DNA or protein will almost invariably detect codominant inheritance, as both alleles are detected. If a mutation produces a phenotype in the heterozygote that is intermediate between the homozygous normal and mutant, the phenotype is referred to as semidominant. A single mutation may confer both a dominant and a recessive phenotype. For example, the mouse patch (Ph) mutation has a heterozygous (dominant) pigmentation phenotype but also a homozygous (recessive) lethal phenotype. As the terms are applied to phenotypes not to genes or alleles, then in the case where a gene has multiple mutant alleles, each can confer a phenotype that is dominant to some, but recessive to other, phenotypes due to other alleles.
Penetrance is a quantitative measure of how often the phenotype occurs in a population; and expressivity is a measure of how strongly a phenotype is expressed in an individual. Particularly in segregating crosses, or where there is a threshold effect on phenotypic manifestation, these measures provide additional ways to describe how particular allelic combinations result in a phenotype.
6.10 Genotype
Genotype is the description of the genetic composition of the animals, usually in terms of particular alleles at particular loci. It may refer to single genes or loci or to many. Genotype can only be determined by assaying phenotype, including test mating to reveal carriers of recessive mutations. Strictly speaking, even direct determination of DNA variants is assaying phenotype not genotype as it is dependent on a particular assay, although it is so close to genotype that it serves as a surrogate.
6.11 Phenotype
Phenotype is the result of interaction between genotype and the environment and can be determined by any assay.
6.12 Quantitative Trait Loci (QTLs)
Quantitative Trait Loci (QTL) are polymorphic loci that contain alleles, which differentially affect the expression of continuously distributed phenotypic traits. Usually these are markers described by statistical association to quantitative variation in the particular phenotypic traits that are controlled by the cumulative action of alleles at multiple loci.
6.13 Haplotype
A haplotype is the association of genetically linked alleles, as found in a gamete. They may be a combination of any type of markers, and may extend over large, genetically separable distances, or be within a short distance such as within a gene and not normally separated.
6.14 Homolog
Genes are homologous if they recognizably have evolved from a common ancestor. Note that genes are either homologous or not; there are no degrees of homology! For example, all globin genes, and myoglobin, are homologs, even though some are more closely related to each other than others. When a measure of relatedness between sequences is required, percent identity or similarity should be used.
6.15 Ortholog
Genes in different species are orthologs if they have evolved from a single common ancestral gene. For example, the beta globin genes of mouse, rat and human are orthologs. Note that several genes in the mouse or rat may have a single ortholog in another species and vice versa.
6.16 Paralog
Paralogous genes are genes within the same species that have arisen from a common ancestor by duplication and subsequent divergence. For example, the mouse alpha globin and beta globin genes are paralogs.
Bestor TH. Transposons reanimated in mice. 2005. Cell 122:322-325.
Committee on Rat Nomenclature, Cochairmen Gill T.J. III, Nomura T. 1992. Definition, nomenclature, and conservation of rat strains. ILAR News 34:S1-S56.
Committee on Standardized Genetic Nomenclature for Mice. 1963. A revision of the standardized genetic nomenclature for mice. J. Hered. 54:159-162.
Committee on Standardized Genetic Nomenclature for Mice. 1973. Guidelines for nomenclature of genetically determined biochemical variants in the house mouse, Mus musculus. Biochem. Genet. 9:369-374.
Committee on Standardized Genetic Nomenclature for Mice, Chair: Lyon, M.F. 1981. Rules and guidelines for gene nomenclature. In: Genetic Variants and Strains of the Laboratory Mouse, Green, M.C. (ed.), First Edition, Gustav Fischer Verlag, Stuttgart, pp. 1-7.
Committee on Standardized Genetic Nomenclature for Mice, Chair: Lyon, M.F. 1989. Rules and guidelines for gene nomenclature. In: Genetic Variants and Strains of the Laboratory Mouse, Lyon, M.F., A.G. Searle (eds.), Second Edition, Oxford University Press, Oxford, pp. 1-11.
Committee on Standardized Genetic Nomenclature for Mice, Chairperson: Davisson, M.T. 1996. Rules and guidelines for gene nomenclature. In: Genetic Variants and Strains of the Laboratory Mouse, Lyon, M.F., Rastan, S., Brown, S.D.M. (eds.), Third Edition, Volume 1, Oxford University Press, Oxford, pp. 1-16.
Ding S, Wu X, Li G, Han M, Zhuang Y, Xu. T. 2005. Efficient transposition of the piggyBac (PB) transposon in mammalian cells and mice. Cell 122:473-483.
Dunn, L.C., H. Gruneberg, G.D. Snell. 1940. Report of the committee on mouse genetics nomenclature. J. Hered. 31:505-506.
Dupuy AJ, Akagi K, Largaespada DA, Copeland NG, Jenkins NA. 2005. Mammalian mutagenesis using a highly mobile somatic Sleeping Beauty transposon system. Nature 436:221-226.
Eppig, JT. 2006. Mouse Strain and Genetic Nomenclature: an Abbreviated Guide. In: Fox J, Barthold S, Davvison M, Newcomer C, Quimby F, Smith A (eds) The Mouse in Biomedical Research, Volume 1, Second Edition. Academic Press. pp.79-98.
International Committee on Standardized Genetic Nomenclature for Mice, Chairperson: Davisson, M.T. 1994. Rules and guidelines for genetic nomenclature in mice. Mouse Genome 92 vii-xxxii.
Levan G., H.J. Hedrich, E.F. Remmers, T. Serikawa, M.C. Yoshida. 1995. Standardized rat genetic nomenclature. Mamm. Genome 6:447-448.
![]()



 nomenclature.exe
nomenclature.exe
















 small_world_bryson.pdf
small_world_bryson.pdf